<전체 실험 개요>
A. FBXO38 gene이 있는 plasmid를 가진 E.coli를 고체LB배지에 배양해서 single colony를 얻는다.
B. Single colony를 따서 액체LB배지에 접종하고 배양한다.
C. 배양액에서 plasmid만 추출해 얻는다.(midi prep)
D. Plasmid의 FBXO38 gene을 다른 vector에 옮긴다.(subcloning)
E. FBXO38 gene을 옮겨준 새 vector를 cell에 transfection 시킨다.
8월 18일
Subcloning하기 전 FBXO38 gene을 PCR을 통해 증폭시켜줘야한다. PCR을 하기 위해선 primer가 필요한데 primer를 어떻게 만드느냐에 따라 PCR의 효율이 달라진다고 한다. 그래서 오늘은 primer를 짜는 방법을 조금 배웠다.
우선 primer는 이런식으로 만들 것이다.

PCR의 primer에 restriction enzyme site 서열을 포함해 PCR산물이 RE site를 갖게 할 것이다. 그러면 새 vector(=destination vector)를 똑같은 RE로 처리한 뒤에 우리가 cloning하려는 gene이 들어갈 수 있을 것이다. 이것이 제대로 성공하기 위해선 target gene(=FBXO38)안에는 우리가 사용하고자 하는 RE site가 없어야 한다. (만약에 target gene안에 그 RE site가 존재한다면 cloning할 때 destination vector뿐만이 아니라 target gene마저 RE에 의해 싹둑싹둑 잘려버리고 만다) Destination vector에는 여러 RE site가 있으므로 이런 조건이 맞는 RE site를 찾아주면 된다.
이런 이유로 primer에는 RE site 서열과 FBXO38 서열 조금이 필요할 것이다.
1. NCBI와 BLAST에서 FBXO38 CDS 찾기
우선 NCBI에서 target gene의 CDS에 대한 정보를 찾는다.
NCBI: https://www.ncbi.nlm.nih.gov/

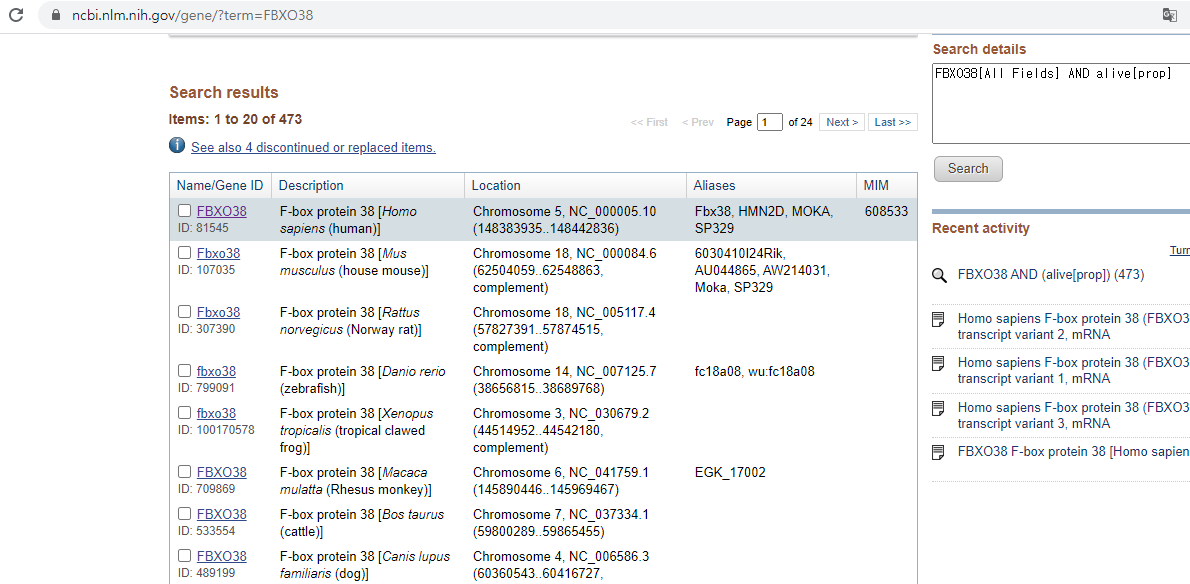



NCBI를 검색한 후 각 isoform에 대한 CDS정보를 찾았으면 BLAST에서 isoform끼리 CDS를 비교해본다.
어떤 isoform을 primer design할 때 사용할지 판단하기 위함이다.
BLAST: https://blast.ncbi.nlm.nih.gov/Blast.cgi




BLAST에서 비교한 결과 isoform a와 b는 일치하고, a와 c는 서열의 앞뒤는 거의 일치하지만 c가 a에 비해 중간부분이 비어있다. Primer를 짤 땐 앞뒤가 이정도 일치하면 큰 문제는 없지만, 일반적으론 서열의 길이가 더 긴 것을 기준으로 primer를 짠다고 한다. 따라서 isoform a를 기준으로 잡을 것이다.
2. NEBcutter
이젠 isoform a의 FBXO38에 어떤 RE site 서열이 존재하는지, 또 cloning을 위해 어떤 RE를 사용하면 될 것인지 확인할 것이다. 이것을 찾아주는 사이트가 NEBcutter이다.
NEBcutter: http://nc2.neb.com/NEBcutter2/index.php


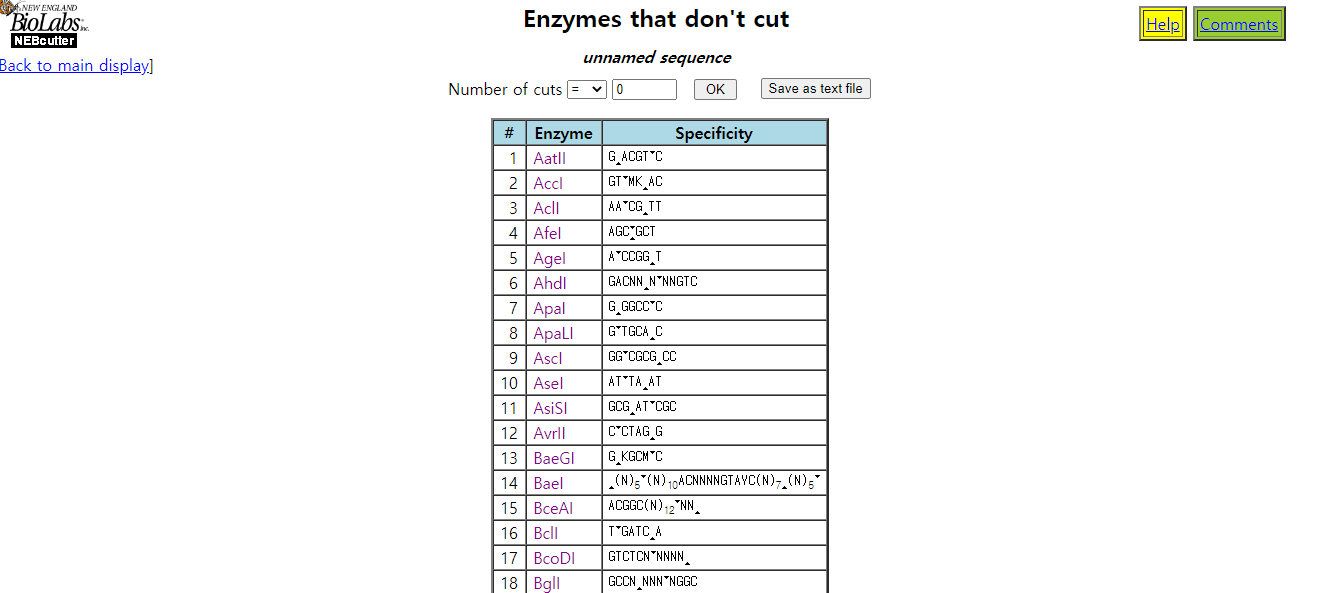
내가 사용해야될 destination vector에는 Hind III, EcoR I, EcoR V, Not I, Xho I, Xba I, Apa I RE site가 존재한다. 이 중 target gene(FBXO38)은 자르지 않는, 즉 위의 표에 제시해준 RE를 찾아야한다. Not I와 Apa I가 그 조건에 부합하는 RE이다.
안타깝게도 우리 랩실에 ApaI이 없다고 한다... 주문시키면 도착하는데 일주일이 걸린다고 한다. 사수 선배가 머리싸맨다...
'실험 protocol' 카테고리의 다른 글
| 200826 PCR/agarose gel/gel electrophoresis/DNA purify (0) | 2020.08.30 |
|---|---|
| 200826 pcr하기 (0) | 2020.08.26 |
| 200813 DNA concentration 구하기 (0) | 2020.08.13 |
| 200812 Plasmid 추출하기(midi prep) (1) | 2020.08.13 |
| 200811 액체LB만들기/접종하기 (0) | 2020.08.13 |